以下为来自各位老师的KOD FX使用例,希望能对大家的实验有所帮助。本实验探讨以用酶处理后的酵母及酵母菌落直接作为样品的PCR。
『Zymolyase处理』 |
【基因名】
将Xanthophyllomyces dendrorous克隆后的产物在质粒状态下导入到
Saccharomyces cerevisiae Y187的序列4种(A-D)
【样品】
用牙签取S. cerevisiae Y187的菌落少许,
在Zymolyase溶液[Zymolyase 2.5 mg/ml、10 mM磷酸钠缓冲液pH7.5、
1.2 M山梨醇(sorbitol)]10 μl中悬浊,取37℃・30分孵育后的溶液1 μl,作为模板添加到 PCR反应液中(total 20 μl)。
【目的片段长】
约3000 bp
【引物条件】
Forward 22 mer 5’-ctattcgatgatgaagataccc-3’
Reverse 19 mer 5’-aaattgagatggtgcacga-3’
【循环】
KOD-FX: 94℃ 4 min
(98℃ 10 sec,60℃ 30 sec, 68 ℃ 4 min)×30cycles
4℃ ∞
※其他公司酶使用其说明书推荐的条件(30cycles)。
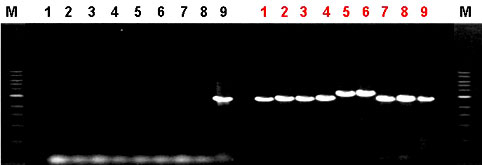
M: 1kb DNA Ladder
公司酶
1~9: KOD FX
1: 导入了带有A序列质粒的菌落1
2: 导入了带有A序列质粒的菌落2
3: 导入了带有B序列质粒的菌落1
4: 导入了带有B序列质粒的菌落2
5: 导入了带有C序列质粒的菌落1
6: 导入了带有C序列质粒的菌落2
7: 导入了带有D序列质粒的菌落1
8: 导入了带有D序列质粒的菌落2
9: 带有B序列的质粒(从酵母中抽提・纯化)(Positive Control)
由于上述实验取得了良好的结果,因此下面继续进行菌落直接PCR(不进行Zymolyase处理)。
『菌落直接法(不进行Zymolyase处理)』 |
【基因名】
其他种类酵母的cDNA文库
【样品】
将导入上述cDNA文库的S. cerevisiae Y187直接添加到PCR反应液中进行悬浊。
【目的片段长】
约500~4000 bp
【引物条件】
Forward 22 mer 5’-ctattcgatgatgaagataccc-3’
Reverse 19 mer 5’-aaattgagatggtgcacga-3’
【循环】
KOD FX: 94℃ 4 min
(98℃ 10 sec,60℃ 30 sec, 68 ℃ 4 min)×30cycles
4℃ ∞

M: 1kb DNA Ladder
1~21: KOD FX
【数据提供】
奈良先端科学技术大学研究生学院 细胞技能学教研室
浮边 健 老师、大津 严生 老师、高木 博史 老师
【来自老师的评语】
以前我们也用过其他公司的Polymerase对酵母进行菌落PCR,但条件稍有差异(菌落的年龄、使用菌体的量等),即会出现有时可扩增,有时无法扩增的情况,很不稳定。
而自从使用『KOD FX』以来,已经进行了100多次菌落PCR,还没有出现过失败的扩增例,且无需用Zymolyase对酵母菌体进行处理(和大肠杆菌的菌落PCR同样)直接将菌体添加到PCR溶液中也能顺利地扩增。
















