用DGGE法对沿海生息的植物-浮游生物(隐藻cryptophyta)的多样性分析 |
【基因名】PsbA(光化学体系II反应中心的D1蛋白 )
特别是以隐藻(Cryptophyta)为目的片段
【样品】海水性过滤膜(Millipore公司的Durapora)过滤的样品
【样品配制方法】
在过滤后的过滤膜里添加溶解液1ml、55℃条件下处理1小时
→去除过滤膜、离心(10,000×g, 10分钟)处理
→上清1~1.5μl作为模板使用
●溶解液组成:20mM Tris-HCl(pH8.0), 5mM EDTA, 0.3% SDS, Proteinase K 200μg/ml |
【片段长】约350bp
【引物序列】
Primer F:5'-CGCCCGCCGCGCCCCGCGCCCGTCCCGCCGCCC
CCGCCCCATGCGTCCTTGGATTTCTGT -3' (60mer)
Primer R:5'-CCARATACCTACAACTGGCCATA-3' (23mer)
【PCR条件】
PCR grade water 3.3μl
2×PCR buffer for KOD FX 15μl
2mM dNTPs 6μl
Primer F (5pmol/μl) 1.8μl
Primer R (5pmol/μl) 1.8μl
KOD FX (1.0U/ ml) 0.6μl
样品 1.5μl
-----------------------------------------------------------------
Total volume 30μl
【PCR循环】
94℃ 2 min
(98℃ 10 sec, 55℃ 30 sec, 68℃ 10 sec)×35cycles
其他公司产品按推荐的条件。
【结果】
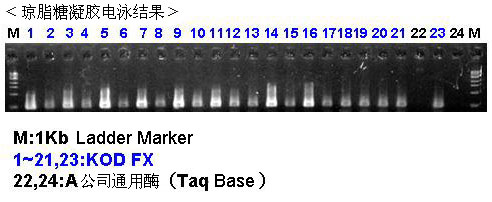
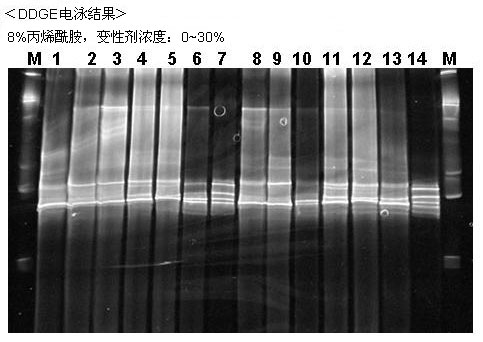
【数据提供】
水产综合研究中心 东北区水产研究所
奥村 裕 老师
【来自老师的评语】
通常用A公司通用型酶(Taq Base)总是不能很顺利地进行PCR,测序时,使用A公司通用型酶 + B公司粗样品用PCR添加剂反应后再进行TA克隆。由于PCR添加剂粘性很高,用琼脂糖凝胶电泳确认没有问题,但如果PCR产物未经纯化用于DGGE时条带就会出现弥散,改变凝胶浓度、变性剂浓度也无法得到改善。
而使用KOD FX,无需抽提DNA即可进行PCR,且能顺利进行DGGE。看了公司网页上所述,使用ProK、SDS后的样品需要纯化,但不纯化直接PCR也能得到很好的结果。循环数一开始尝试了30个循环,但35个循环更好。此外,开始很担心GC发卡结构的引物能否顺利扩增,最终证明也没有问题,顺利地进行了PCR。








